Notícia
Nova ferramenta prevê mudanças que podem tornar as variantes da COVID-19 mais infecciosas
Ferramenta pode permitir a vigilância computacional do SARS-CoV-2 e fornecer um aviso prévio de variantes potencialmente perigosas com um potencial de afinidade de ligação ainda maior
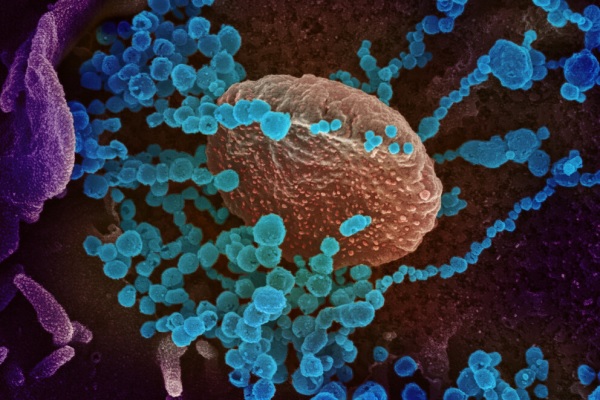
NIAID/NIH
Fonte
Penn State | Universidade Estadual da Pensilvânia
Data
segunda-feira, 4 outubro 2021 06:10
Áreas
Inteligência Artificial. Saúde Pública.
À medida que o SARS-CoV-2 continua a evoluir, podem surgir novas variantes com capacidade aumentada de infectar e escapar do sistema imunológico dos hospedeiros. A primeira etapa chave na infecção é quando a proteína spike do vírus se liga ao receptor ACE2 nas células humanas. Pesquisadores da Universidade Estadual da Pensilvânia (Penn State), nos Estados Unidos, criaram uma nova estrutura que pode prever com precisão razoável as mudanças de aminoácidos na proteína spike do vírus que pode melhorar sua ligação às células humanas e tornar o vírus mais infeccioso.
A ferramenta pode permitir a vigilância computacional do SARS-CoV-2 e fornecer um aviso prévio de variantes potencialmente perigosas com um potencial de afinidade de ligação ainda maior. Isso pode ajudar na implementação precoce de medidas de saúde pública para prevenir a propagação do vírus e talvez até mesmo pode informar as formulações de reforço de vacinas.
“Variantes emergentes podem ser altamente contagiosas em humanos e outros animais”, disse o Dr. Suresh Kuchipudi, professor de Ciências Veterinárias e Biomédicas e diretor do Animal Diagnostic Lab da Penn State. “Portanto, é fundamental avaliar de forma proativa quais mudanças de aminoácidos provavelmente podem aumentar a infecciosidade do vírus. Nossa estrutura é uma ferramenta poderosa para determinar o impacto das alterações de aminoácidos na proteína spike do SARS-CoV-2 que afetam a capacidade do vírus de se ligar aos receptores ACE2 em humanos e em várias espécies animais”, continuou o professor.
A equipe usou um novo procedimento computacional de duas etapas para criar um modelo para prever quais mudanças nos aminoácidos podem ocorrer no domínio de ligação ao receptor (RBD) da proteína spike do SARS-CoV-2 que poderiam afetar sua capacidade de se ligar aos receptores ACE2 de células humanas e de outras células animais.
De acordo com o Dr. Suresh Kuchipudi, as variantes atualmente em circulação incluem uma ou mais mutações que levaram a alterações de aminoácidos no RBD da proteína spike.
“Essas mudanças de aminoácidos podem ter conferido vantagens de aptidão e aumento da infectividade por meio de uma variedade de mecanismos. O aumento da afinidade de ligação do RBD da proteína spike com o receptor ACE2 humano é um desses mecanismos”, explicou o pesquisador.
O professor Kuchipudi explicou que a ligação da proteína spike ao receptor ACE2 é a primeira e crucial etapa na entrada do vírus na célula: “A força de ligação entre RBD e ACE2 afeta diretamente a dinâmica da infecção e potencialmente a progressão da doença. A capacidade de prever com segurança os efeitos das alterações de aminoácidos do vírus na capacidade de seu RBD de interagir mais fortemente com o receptor ACE2 pode ajudar a avaliar as implicações para a saúde pública e o potencial de transbordamento e adaptação em humanos e outros animais”.
O Dr. Costas Maranas, professor do Departamento de Engenharia Química da Penn State, liderou o desenvolvimento do novo procedimento de duas etapas da equipe. Primeiro, os pesquisadores testaram o poder preditivo de uma técnica de mecânica molecular chamada MM-GBSA, para quantificar a afinidade de ligação do RBD para ACE2. A análise de MM-GBSA resume vários tipos de contribuições de energia associadas ao RBD do vírus ‘grudado’ ao receptor ACE2 humano. Usando dados de variantes já existentes, a equipe descobriu que esta técnica era apenas parcialmente capaz de prever a afinidade de ligação do RBD do SARS-CoV-2 para ACE2.
Portanto, o Dr. Costas Maranas e a equipe de pesquisadores exploraram o uso dos termos de energia da análise MM-GBSA como recursos em um modelo de regressão de rede neural – um tipo de algoritmo de aprendizagem profunda – e treinaram o modelo usando dados experimentalmente disponíveis sobre ligação em variantes com alterações em um único aminoácido. Eles descobriram que podiam prever com mais de 80% de precisão se certas alterações de aminoácidos melhoraram ou pioraram a afinidade de ligação para o conjunto de dados explorado.
“Este [método] MM-GBSA combinado com uma abordagem de modelo de rede neural parece ser bastante eficaz em prever o efeito das alterações de aminoácidos não utilizadas durante o treinamento do modelo”, disse o Dr. Maranas.
O modelo também permitiu a previsão da força de ligação de várias alterações de aminoácidos do SARS-CoV-2 já observadas nas variantes Alfa, Beta, Gama e Delta. Isso pode fornecer os meios computacionais para prever tais afinidades em variantes ainda a serem descobertas. No entanto, embora a ferramenta computacional possa encontrar mudanças de aminoácidos que aumentem ainda mais a afinidade de ligação, estas mudanças ainda não foram observadas em variantes circulantes. Isso pode significar que tais mudanças podem interferir com outros requisitos de infecção viral produtiva. É um lembrete de que a ligação com o receptor ACE2 não é tudo.
Os resultados foram publicados na revista científica Proceedings of the National Academy of Sciences (PNAS).
Acesse o artigo científico completo (em inglês).
Acesse a notícia completa na página da Universidade Estadual da Pensilvânia (em inglês).
Fonte: Sara LaJeunesse, Universidade Estadual da Pensilvânia. Imagem: Observação de microscópio eletrônico de varredura mostra o vírus SARS-CoV-2 (objetos azuis redondos) emergindo da superfície de células cultivadas em laboratório. O vírus mostrado foi isolado de um paciente nos EUA. Fonte: NIAID/NIH.
Em suas publicações, o Portal Tech4Health da Rede T4H tem o único objetivo de divulgação científica, tecnológica ou de informações comerciais para disseminar conhecimento. Nenhuma publicação do Portal Tech4Health tem o objetivo de aconselhamento, diagnóstico, tratamento médico ou de substituição de qualquer profissional da área da saúde. Consulte sempre um profissional de saúde qualificado para a devida orientação, medicação ou tratamento, que seja compatível com suas necessidades específicas.
Os comentários constituem um espaço importante para a livre manifestação dos usuários, desde que cadastrados no Portal Tech4Health e que respeitem os Termos e Condições de Uso. Portanto, cada comentário é de responsabilidade exclusiva do usuário que o assina, não representando a opinião do Portal Tech4Health, que pode retirar, sem prévio aviso, comentários postados que não estejam de acordo com estas regras.




Apenas usuários cadastrados no Portal tech4health t4h podem comentar, Cadastre-se! Por favor, faça Login para comentar